Genetischer Code
Als genetischer Code wird die Weise bezeichnet, mit der die Nukleotidsequenz eines RNA-Einzelstrangs in die Aminosäurensequenz der Polypeptidkette eines Proteins übersetzt wird. In der Zelle geschieht dies, nachdem zuvor die in der Abfolge von Basenpaaren des DNA-Doppelstrangs niedergelegte Erbinformation in die Sequenz des RNA-Einzelstrangs (Boten- oder Messenger-Ribonukleinsäure, mRNA) umgeschrieben wurde.
Dieser genetische Code ist bei allen bekannten Arten von Lebewesen in den Grundzügen gleich. Er ordnet einem Triplett von drei aufeinanderfolgenden Nukleobasen der Nukleinsäuren – dem sogenannten Codon – jeweils eine bestimmte proteinogene Aminosäure zu. Die Übersetzung, Translation genannt, findet an den Ribosomen im Zytosol einer Zelle statt. Sie bilden nach Vorgabe der Sequenz von Nukleotiden einer mRNA die Sequenz von Aminosäuren eines Peptids, indem jedem Codon über das Anticodon einer Transfer-Ribonukleinsäure (tRNA) eine bestimmte Aminosäure zugewiesen und diese mit der vorherigen verbunden wird. Auf diese Weise wird eine bestimmte vorgegebene Information in die Form einer Peptidkette überführt, die sich dann zur besonderen Form eines Proteins faltet.
Je komplexer Lebewesen jedoch sind, desto höher scheint der Anteil genetischer Information zu sein, der nicht in Proteine übersetzt wird. Ein beträchtlicher Teil an nicht-codierender DNA wird zwar in RNAs transkribiert, aber nicht per Translation in eine Peptidkette übersetzt. Zu diesen nicht für Protein codierenden RNA-Spezies des Transkriptoms gehören neben den für die Translation erforderlichen tRNAs und ribosomalen RNAs (rRNA) eine Reihe weiterer, meist kleiner RNA-Formen. Diese dienen in vielfältiger Weise der Regulation verschiedener zellulärer Prozesse – so der Transkription selbst, wie auch der möglichen Translation, außerdem einer eventuellen DNA-Reparatur, und darüber hinaus besonderen epigenetischen Markierungen von DNA-Abschnitten sowie u. a. verschiedenen Funktionen des Immunsystems.

GCC passt.Die Transfer-Ribonukleinsäuren, tRNAs, enthalten an prominenter Stelle einer Schleife des kleeblattähnlichen Moleküls ein kennzeichnendes Nukleotid-Triplett, das sie voneinander unterscheidet. Es besteht jeweils aus drei Nukleotiden, die den Nukleotiden eines bestimmten Codons entsprechen, indem sie komplementär zu diesen sind und so ein dreigliedriges Anticodon bilden. Codon und Anticodon passen basenpaarend zueinander und ihnen ist die gleiche spezifische Aminosäure zugeordnet. Eine tRNA wird jeweils mit derjenigen Aminosäure beladen, für die das zu ihrem Anticodon passende Codon steht. Auf diese Weise, durch die spezifische Bindung einer Aminosäure an eine tRNA mit einem bestimmten Anticodon, wird also das Zeichen für eine bestimmte Aminosäure, das Codon, in die genetisch codierte Aminosäure übersetzt.
Streng genommen ist der genetische Code also schon in der Struktur der verschiedenen tRNA-Arten enthalten: Denn ein jedes tRNA-Molekül enthält eine derart strukturierte Aminosäure-Bindungsstelle, dass daran nur jene Aminosäure gebunden wird, die seinem Anticodon nach dem genetischen Code entspricht. Nach Bindung an ihre tRNA steht eine Aminosäure für die Biosynthese von Proteinen am Ribosom zur Verfügung, sodass sie als nächstes Glied der Polypeptidkette angefügt werden kann – falls das Anticodon der tRNA zu einem Codon in der vorgegebenen Nukleotidsequenz der mRNA passt.
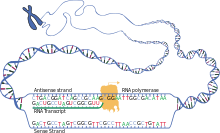
Als Voraussetzung für diese Proteinsynthese muss der DNA-Abschnitt eines Gens zunächst in eine Ribonukleinsäure (RNA) umgeschrieben werden (Transkription). Dabei können in eukaryoten Zellen bestimmte Teile dieser hnRNA gezielt entfernt (Spleißen) oder danach verändert werden (RNA-Editing); anschließend wird diese vorläufige prä-mRNA weiter prozessiert zur definitiven mRNA, die schließlich aus dem Zellkern exportiert wird. Denn erst an den Ribosomen, die frei im Zytosol vorliegen können oder an das endoplasmatische Reticulum gebunden sind, werden anhand der mRNA-Vorlage dann die Aminosäuren der zu den Codons passenden tRNAs miteinander zu einem Polypeptid verknüpft.
Dieser Vorgang, mit dem die Information eines Gens in der Form eines Proteins ausgedrückt wird (Genexpression), ergibt sich somit aus einer Folge von Schritten. Hierbei werden die Hauptprozesse unterschieden als (1) Transkription – ein Abschnitt der DNA des Genoms wird durch RNA-Polymerase in RNA umgeschrieben – und (2) posttranskriptionale Modifikation – eine RNA des Transkriptoms wird verändert – sowie (3) Translation – eine mRNA wird am Ribosom in ein Polypeptid übersetzt. Daran kann sich (4) noch eine posttranslationale Modifikation anschließen – ein Polypeptid des Proteoms wird verändert. Im Ablauf dieser Prozesse bis hin zur Bereitstellung eines funktionstragenden Proteins ist die Translation also der Schritt, in dem die genetische Information der Basentriplett-Abfolge in eine Aminosäure-Abfolge umgesetzt wird.
Die eigentliche Anwendung des genetischen Codes, nämlich die Übersetzung einer Nukleotidsequenz in eine Aminosäure anhand des Codons beziehungsweise des Anticodons, findet schon bei der Bindung einer Aminosäure an ihre tRNA durch die jeweilige Aminoacyl-tRNA-Synthetase statt, also bei der Vorbereitung der Aminosäuren für ihren möglichen Zusammenbau in einem Protein. Einige wenige Basentripletts codieren nicht für eine Aminosäure. Insofern sie in diesem Sinn keine Bedeutung tragen, werden sie auch Nonsens-Codons genannt; diese führen bei der Translation zu einem Stop, der die Proteinsynthese beendet, und heißen daher auch Stopcodons.
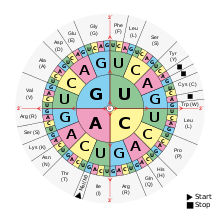
Alle Lebewesen benutzen in Grundzügen denselben genetischen Code. Die wohl am häufigsten gebrauchte Version ist in den folgenden Tabellen angegeben. Sie zeigen für diesen Standard-Code, welche Aminosäuren von einem der 43 = 64 möglichen Codons gemeinhin codiert werden, bzw. welches Codon in eine der 20 kanonischen Aminosäuren übersetzt wird. So steht zum Beispiel das Codon GAU für die Aminosäure Asparaginsäure (Asp), und Cystein (Cys) wird von den Codons UGU und UGC codiert. Die in der Tabelle angegebenen Basen sind Adenin (A), Guanin (G), Cytosin (C) und Uracil (U) der Ribonukleotide der mRNA; in den Nukleotiden der DNA tritt dagegen Thymin (T) anstelle von Uracil auf. Bei der Transkription eines DNA-Abschnitts dient einer RNA-Polymerase der codogene Strang als Matrize für das Transkript: die DNA-Basensequenz wird basenpaarend in die komplementäre RNA-Basensequenz umgeschrieben beim Aufbau eines RNA-Strangs. Damit wird auf die in DNA vererbbar abgelegte genetische Information zugegriffen, die dann in mRNA für die Proteinbiosynthese zur Verfügung steht.
Geschichte der Entdeckung
[Bearbeiten | Quelltext bearbeiten]Der erste Vergleich von großen, aus zahlreichen Bausteinen bestehenden Erbmolekülen mit einer Schrift stammt von Friedrich Miescher, dem Entdecker der Nukleinsäuren, wie aus den erst posthum 1897 veröffentlichten Briefen an den Mediziner Wilhelm His, seinen Onkel, hervorgeht. Es sei völlig überflüssig, aus Eizelle und Spermazelle „eine Vorratskammer zahlloser chemischer Stoffe zu machen, deren jeder Träger einer besonderen erblichen Eigenschaft sein soll,“ schrieb er 1892.[1] Weder das Protoplasma noch der Kern der Zelle bestünden aus zahllosen chemischen Stoffen, sondern vielmehr aus „ganz wenigen chemischen Individuen, von allerdings vielleicht sehr kompliziertem chemischen Bau.“[1] Der Schlüssel zu Vererbung und zur Sexualität liegt nach diesen Überlegungen Mieschers in der Stereochemie von Großmolekülen. Die Größe und Kompliziertheit der beteiligten chemischen Bauformen erlaube eine kolossale Menge von Stereoisomerien, sodass „aller Reichtum und alle Mannigfaltigkeit erblicher Übertragungen ebenso gut darin ihren Ausdruck finden können, als die Worte und Begriffe aller Sprachen in den 24–30 Buchstaben des Alphabets.“[1]
Albrecht Kossel, der Mieschers Arbeiten an Nukleinsäuren fortgeführt und bereits 1891 als deren Spaltprodukte die Nukleinbasen entdeckt hatte, vertiefte diesen heuristischen Schriftvergleich der Erbinformation in seiner Harvey Lecture „The chemical composition of the cell“ von 1911.[2] Diesen Vergleich griff Max Planck in seinem Vortrag Positivismus und reale Außenwelt (1930) auf und später Erwin Schrödinger im Dubliner Exil in Vorträgen und der wirkmächtigen Schrift What is Life? (1944) für die Frage, wie genetische Information in Molekülen gespeichert und weitergegeben werden kann. Der Philosoph Hans Blumenberg weist in seinem Werk Die Lesbarkeit der Welt (1986) in der Episode Der genetische Code und seine Leser darauf hin, dass die späten wichtigen Äußerungen des bereits schwer erkrankten Friedrich Miescher in der Biologie kaum rezipiert sind.[3]
All diese Spekulationen erhielten allerdings erst eine feste Basis, als die Molekularbiologie weiter fortschritt. In den 1940er Jahren gelang Oswald Avery der Nachweis, dass die DNA Träger der Erbinformation ist. 1953 klärten James Watson und Francis Crick deren Struktur und wiesen auf einen möglichen Mechanismus der Vervielfältigung hin. George Gamow gab Francis Crick den ersten Anstoß zur Aufklärung des genetischen Codes, als er ihm in einem Brief mitteilte, dass für die Kodierung von 20 Aminosäuren mindestens Dreier-Kombinationen (Tripletts) der vier Basen der DNS nötig sind (da mit Zweierkombinationen nur Aminosäuren kodierbar sind).[4] Gamow veröffentlichte dies auch in einem Brief an Nature 1954[5] und in den Mitteilungen der dänischen Akademie der Wissenschaften.[6]
In der ersten Hälfte der 1960er Jahre herrschte unter Biochemikern eine gewisse Konkurrenz um das Verständnis des genetischen Codes. Die grundlegende Idee, dass die Aminosäuren durch Basentripletts kodiert werden, konnte 1961 eine Gruppe um Francis Crick und Sydney Brenner bestätigen.[7] Den ersten Schritt zur Aufklärung der Triplett-Codewörter für bestimmte Aminosäuren machte Marshall Nirenbergs Labor an den National Institutes of Health in Bethesda. Am 27. Mai 1961 in der Zeit von 3 Uhr bis 8 Uhr morgens[8] gelang dem deutschen Biochemiker Heinrich Matthaei, damals Post-Doktorand, im Rahmen des Poly-U-Experiments von Nirenberg und Matthaei der entscheidende Durchbruch: die Entschlüsselung des Codons UUU für die Aminosäure Phenylalanin. Nirenberg trug darüber im August 1961 auf dem Internationalen Biochemie-Kongress in Moskau vor, ohne zunächst viel Aufsehen zu erregen, da er kaum bekannt war. Erst die von Crick angeregte Wiederholung des Vortrags auf dem Kongress elektrisierte das Publikum; das Experiment zählt zu den bedeutendsten in der Genetik des 20. Jahrhunderts. Um die Entschlüsselung der übrigen Codons entbrannte danach ein Wettkampf, insbesondere zwischen den Gruppen um Nirenberg und um Severo Ochoa. 1966 waren alle 64 Basentripletts untersucht und für nahezu alle Codons entsprechende Aminosäuren herausgefunden. Dabei erwies sich, dass einige besondere Codons, denen keine Aminosäure zugeordnet werden konnte, zwar in diesem Sinn keine Bedeutung tragen (Nonsense-Codon), aber eine wichtige Funktion erfüllen, indem sie ein Stopsignal darstellen (Stopcodon).
Codon
[Bearbeiten | Quelltext bearbeiten]Genetische Information für den Aufbau von Proteinen ist in bestimmten Abschnitten der Basensequenz von Nukleinsäuren enthalten. Von DNA in RNA umgeschrieben (transkribiert), wird sie für die Biosynthese von Proteinen verfügbar. Die im offenen Leserahmen vorliegende Basensequenz wird am Ribosom abgelesen und nach dem genetischen Code übersetzt (translatiert) in die Aminosäurensequenz der synthetisierten Peptidkette, die Primärstruktur eines Proteins. Dabei wird die Basenfolge schrittweise in Dreiergruppen zerlegt gelesen und jedem dieser Tripletts je eine dazu passende tRNA zugeordnet, beladen mit einer bestimmten Aminosäure. Die Aminosäure wird jeweils durch Peptidbindung an die vorherige gebunden. Auf diese Weise codiert der Sequenzabschnitt für Protein.
Als Codon bezeichnet man das Variationsmuster einer Abfolge von drei Nukleobasen der mRNA, eines Basentripletts, das für eine Aminosäure codieren kann. Insgesamt existieren 43 = 64 mögliche Codons, davon codieren 61 für die insgesamt 20 kanonischen der proteinogenen Aminosäuren; die restlichen drei sind sogenannte Stopcodons zur Termination der Translation. Diese können unter bestimmten Umständen genutzt werden, zwei weitere nicht-kanonische Aminosäuren zu codieren. Damit gibt es für fast alle der Aminosäuren mehrere verschiedene Codierungen, jeweils meist recht ähnliche. Die Codierung als Triplett ist jedoch insofern notwendig, als bei einer Duplett-Codierung nur 42 = 16 mögliche Codons entstehen würden, womit schon für die zwanzig kanonischen oder Standard-Aminosäuren nicht genügend Möglichkeiten gegeben wären.
| 2. Base | |||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| U | C | A | G | ||||||||||||||||||||||||||||||||||
| 1. Base | U |
|
|
|
| ||||||||||||||||||||||||||||||||
| C |
|
|
|
| |||||||||||||||||||||||||||||||||
| A |
|
|
|
| |||||||||||||||||||||||||||||||||
| G |
|
|
|
| |||||||||||||||||||||||||||||||||
|
* Das Triplett des Codons | ||||||||||||||||||||||||||||||||||||
Die angegebenen Codons gelten für die Nukleotidsequenz einer mRNA. Sie wird in 5′→3′ Richtung am Ribosom abgelesen und übersetzt in die Aminosäurensequenz eines Polypeptids.
| Az | AS | AS | Codon |
|---|---|---|---|
| 1 | Start | > | AUG |
| 1 | Met | M | AUG |
| 1 | Trp | W | UGG |
| 1 | Sec | U | (UGA) |
| 1 | Pyl | O | (UAG) |
| 2 | Tyr | Y | UAU UAC |
| 2 | Phe | F | UUU UUC |
| 2 | Cys | C | UGU UGC |
| 2 | Asn | N | AAU AAC |
| 2 | Asp | D | GAU GAC |
| 2 | Gln | Q | CAA CAG |
| 2 | Glu | E | GAA GAG |
| 2 | His | H | CAU CAC |
| 2 | Lys | K | AAA AAG |
| 3 | Ile | I | AUU AUC AUA |
| 4 | Gly | G | GGU GGC GGA GGG |
| 4 | Ala | A | GCU GCC GCA GCG |
| 4 | Val | V | GUU GUC GUA GUG |
| 4 | Thr | T | ACU ACC ACA ACG |
| 4 | Pro | P | CCU CCC CCA CCG |
| 6 | Leu | L | CUU CUC CUA CUG UUA UUG |
| 6 | Ser | S | UCU UCC UCA UCG AGU AGC |
| 6 | Arg | R | CGU CGC CGA CGG AGA AGG |
| 3 | Stop | < | UAA UAG UGA |
Die Translation beginnt mit einem Start-Codon. Doch sind daneben bestimmte Initiationssequenzen und -faktoren nötig, um die Bindung der mRNA an ein Ribosom herbeizuführen und den Prozess zu starten. Dazu gehört auch eine spezielle Initiator-tRNA, welche die erste Aminosäure trägt. Das wichtigste Start-Codon ist AUG, das für Methionin codiert. Auch können ACG und CUG – sowie GUG und UUG in prokaryoten Zellen – als Startcodon dienen, allerdings mit geringerer Effizienz. Die erste Aminosäure ist aber zumeist ein – bei Bakterien und in Mitochondrien N-fomyliertes – Methionin.[9]
Die Translation endet mit einem der drei Stop-Codons, auch Terminations-Codons genannt. Anfangs wurden diesen Codons auch Namen gegeben – UAG ist amber (bernsteinfarben), UGA ist opal (opalfarben), und UAA ist ochre (ockerfarben) (ein Wortspiel auf den Nachnamen ihres Entdeckers Harris Bernstein).
Während das Codon UGA zumeist als Stop gelesen wird, kann es selten und nur unter bestimmten Bedingungen für eine 21. (proteinogene) Aminosäure stehen: Selenocystein (Sec). Die Biosynthese und der Einbaumechanismus von Selenocystein in Proteine unterscheiden sich stark von dem aller anderen Aminosäuren: seine Insertion erfordert einen neuartigen Translationsschritt, bei dem ein UGA im Rahmen einer bestimmten Sequenzumgebung und zusammen mit bestimmten Cofaktoren anders interpretiert wird. Hierfür ist außerdem eine für Selenocystein bestimmte, strukturell einzigartige tRNA (tRNASec) erforderlich, die bei Vertebraten auch mit zwei chemisch verwandten Aminosäuren beladen werden kann: neben Selenocystein auch Serin oder Phosphoserin.
Einige Archaeen und Bakterien können daneben ein kanonisches Stopcodon UAG auch in eine weitere (22.) proteinogene Aminosäure übersetzen: Pyrrolysin (Pyl). Sie verfügen über eine spezielle tRNAPyl sowie ein spezifisches Enzym, diese zu beladen (Pyrrolysyl-tRNA-Synthetase).
Manche kurze DNA-Sequenzen kommen im Genom einer Art nur selten oder gar nicht vor (Nullomere). Bei Bakterien erweisen sich manche dieser als toxisch; auch das Codon AGA, welches die Aminosäure Arginin codiert, wird in Bakterien vermieden (stattdessen wird CGA verwendet).[10] Es gibt durchaus artspezifische Unterschiede in der Codonverwendung.[11] Unterschiede im Gebrauch von Codons bedeuten nicht unbedingt Unterschiede in der Häufigkeit verwendeter Aminosäuren. Denn für die meisten der Aminosäuren gibt es mehr als ein einziges Codon, wie die obenstehende Tabelle zeigt.
Degeneration und Fehlertoleranz
[Bearbeiten | Quelltext bearbeiten]Soll eine bestimmte Aminosäure codiert werden, kann oft unter mehreren Codons mit gleicher Bedeutung gewählt werden. Der genetische Code ist ein Code, bei dem mehrere Ausdrücke die gleiche Bedeutung haben, dieselbe semantische Einheit also durch unterschiedliche syntaktische Symbole codiert werden kann. Im Vergleich zu einem Codierungssystem, bei dem jeder semantischen Einheit je ein syntaktischer Ausdruck entspricht und umgekehrt, nennt man solch einen Code degeneriert.
Es hat Vorteile, dass für die circa 20 translational einzubauenden Aminosäuren über 60 Codons verfügbar sind. Dargestellt werden sie jeweils als Kombination aus drei Nukleotiden mit je vier möglichen Basen, sodass es 64 Kombinationen gibt. Deren jeweilige Zuordnung zu einer Aminosäure ist so, dass sehr ähnliche Codon-Variationen für eine bestimmte Aminosäure codieren. Durch die Fehlertoleranz des genetischen Codes genügen oft schon zwei Nukleotide, um eine Aminosäure sicher anzugeben.[12]

Die für eine Aminosäure codierenden Basentripletts unterscheiden sich meist in nur einer der drei Basen; sie haben den minimalen Abstand im Coderaum, siehe Hammingdistanz bzw. Levenshtein-Distanz. Meist unterscheiden sich Tripletts in der dritten Base, der „wackelnden“, die bei Translationen am ehesten falsch gelesen wird (siehe „wobble“-Hypothese).[13] Für den Proteinaufbau häufig nötige Aminosäuren werden von mehr Codons repräsentiert als selten gebrauchte. Eine tiefere Analyse des genetischen Codes offenbart weitere Zusammenhänge etwa bezüglich des Molvolumens und des hydrophoben Effekts (siehe Abbildung).
Bemerkenswert ist auch, dass die Base in der Mitte eines Tripletts den Charakter der zugeordneten Aminosäure weitgehend angeben kann: So sind es im Falle von _ U _ hydrophobe, aber hydrophile im Falle von _ A _. Bei _ C _ sind es unpolare oder polare ohne Ladung, solche mit geladenen Seitenketten treten bei _ G _ als auch bei _ A _ auf, mit negativer Ladung nur bei _ A _ (siehe Tabelle oben). Deshalb sind Radikalsubstitutionen – der Tausch gegen Aminosäuren eines anderen Charakters – oft Folge von Mutationen in jener zweiten Position. Mutationen in der dritten Position („wobble“) bewahren dagegen oft als konservative Substitution die jeweilige Aminosäure oder zumindest deren Charakter. Da Transitionen (Umwandlung von Purinen bzw. Pyrimidinen ineinander, beispielsweise C→T) aus mechanistischen Gründen häufiger auftreten als Transversionen (Umwandlung eines Purins in ein Pyrimidin oder umgekehrt; dieser Prozess setzt zumeist eine Depurinierung voraus), ergibt sich eine weitere Erklärung für die konservativen Eigenschaften des Codes.
Entgegen früheren Annahmen ist die erste Codon-Position oft wichtiger als die zweite Position,[14] vermutlich weil allein Änderungen der ersten Position die Ladung umkehren können (von einer positiv geladenen zu einer negativ geladenen Aminosäure oder umgekehrt). Eine Ladungsumkehr aber kann für die Protein-Funktion dramatische Folgen haben. Dies übersah man bei vielen früheren Studien.
Die sogenannte Degeneration der genetischen Codes macht es auch möglich, die genetische Information weniger empfindlich gegenüber äußeren Einwirkungen zu speichern. Dies gilt insbesondere in Bezug auf Punktmutationen, sowohl für synonyme Mutationen (die zur gleichen Aminosäure führen) als auch für nichtsynonyme Mutationen, die zu Aminosäuren mit ähnlichen Eigenschaften führen.[15]
Offenbar war es schon früh in der Evolutionsgeschichte hilfreich, die Anfälligkeit der Codierung gegenüber fehlerhaft gebildeten Codons zu senken. Die Funktion eines Proteins wird durch dessen Struktur bestimmt. Diese hängt von der Primärstruktur ab, der Sequenz der Aminosäuren: wie viele, welche und in welcher Reihenfolge zu einer Peptidkette verknüpft werden. Diese Angaben enthält die Basensequenz als genetische Information. Eine erhöhte Fehlertoleranz der Codierung sichert die richtige Decodierung. Wird bei einer falschen eher eine Aminosäure mit ähnlichem Charakter eingebaut, verändert dies die Protein-Funktion weniger, als wenn es eine ganz anderen Charakters wäre.
Ursprung des genetischen Codes
[Bearbeiten | Quelltext bearbeiten]Die Verwendung des Wortes „Code“ geht auf Erwin Schrödinger zurück, der die Begriffe „hereditary code-script“, „chromosome code“ und „miniature code“ in einer Vortragsreihe 1943 verwendet hatte, die er 1944 zusammenfasste und als Grundlage für sein Buch „Was ist Leben?“ aus dem Jahr 1944 verwendete.[16] Der genaue Sitz oder Träger dieses Codes war zu diesem Zeitpunkt noch unklar.
Früher glaubte man, der genetische Code sei zufällig entstanden. Noch 1968 bezeichnete Francis Crick ihn als „eingefrorenen Zufall“.[17][18] Er ist jedoch das Resultat einer strengen Optimierung hinsichtlich der Fehlertoleranz.[19] Fehler sind besonders gravierend für die räumliche Struktur eines Proteins, wenn sich die Hydrophobie einer fälschlich eingebauten Aminosäure deutlich vom Original unterscheidet. Im Rahmen einer statistischen Analyse erweisen sich in dieser Hinsicht unter einer Million Zufallscodes nur 100 besser als der tatsächliche. Berücksichtigt man bei der Berechnung der Fehlertoleranz zusätzliche Faktoren, die typischen Mustern von Mutationen und Lesefehlern entsprechen, so reduziert sich diese Zahl sogar auf 1 von 1 Million.[20]
Universalität des Codes
[Bearbeiten | Quelltext bearbeiten]Grundprinzip
[Bearbeiten | Quelltext bearbeiten]Bemerkenswert ist, dass der genetische Code für alle Lebewesen im Prinzip gleich ist, alle Lebewesen sich also der gleichen „genetischen Sprache“ bedienen.[21] Nicht nur, dass genetische Information bei allen in der Sequenz von Nukleinsäuren vorliegt, und für den Aufbau von Proteinen immer in Tripletts abgelesen wird. Bis auf wenige Ausnahmen steht auch ein bestimmtes Codon jeweils für dieselbe Aminosäure; den gemeinhin üblichen Gebrauch gibt der Standard-Code wieder. Daher ist es möglich, in der Gentechnik z. B. das Gen für menschliches Insulin in Bakterien einzuschleusen, damit diese dann das Hormonprotein Insulin produzieren. Dieses von allen Organismen geteilte gemeinsame Grundprinzip der Codierung wird als „Universalität des Codes“ bezeichnet. Es erklärt sich aus der Evolution so, dass der genetische Code schon sehr früh in der Entwicklungsgeschichte des Lebens ausgestaltet und dann von allen sich entwickelnden Arten weitergegeben wurde. Eine solche Generalisierung schließt nicht aus, dass sich die Häufigkeit verschiedener Codewörter zwischen den Organismen unterscheiden kann (siehe Codon Usage).
Varianten
[Bearbeiten | Quelltext bearbeiten]Daneben gibt es aber auch verschiedene Varianten, die vom Standard-Code abweichen, bei denen also einige wenige Codons in eine andere als die in der #Standard-Codon-Tabelle angegebene Aminosäure übersetzt werden. Manche dieser Abweichungen lassen sich taxonomisch eingrenzen, sodass besondere Codes definiert werden können. Derart werden inzwischen schon über dreißig variante genetische Codes unterschieden.[22]
Bei eukaryoten Zellen zeigen jene Organellen, die über ein eigenständiges genomisches System verfügen und vermutlich von symbiotischen Bakterien abstammen (Endosymbionten-Theorie), eigene Varianten des genetischen Codes. In Mitochondrien sind so für deren eigene DNA (mtDNA, Mitogenom syn. Chondriom) über zehn abgewandelte Formen mitochondrialen Codes bekannt. Diese weichen jeweils ab vom nukleären Code für die Erbsubstanz im Kern, das Kern-Genom (Karyom). Daneben haben die in Pflanzenzellen zusätzlich vorkommenden Plastiden einen eigenen Code für ihre plastidäre DNA (cpDNA, Plastom).
Auch die Wimpertierchen (Ciliophora) zeigen Abweichungen vom Standard-Code: UAG, nicht selten auch UAA, codieren für Glutamin; diese Abweichung findet sich auch in einigen Grünalgen. UGA steht auch manchmal für Cystein. Eine weitere Variante findet sich in der Hefe Candida, wo CUG Serin codiert.
Des Weiteren gibt es einige Varianten von Aminosäuren, die nicht nur von Bakterien (Bacteria) und Archaeen (Archaea) während der Translation durch Recodierung eingebaut werden können; so kann UGA, wie oben beschrieben, Selenocystein und UAG Pyrrolysin codieren, im Standard-Code beidenfalls Stop-Codons.
Darüber hinaus sind noch weitere Abweichungen vom Standard-Code bekannt, die oft die Initiation (Start) oder die Termination (Stop) betreffen; insbesondere in Mitochondrien ist einem Codon (Basentriplett der mRNA) öfters nicht die übliche Aminosäure zugeordnet. In der folgenden Tabelle sind einige Beispiele aufgeführt:
| Vorkommen | Codon | Standard | Abweichung |
|---|---|---|---|
| Mitochondrien (bei allen bis jetzt untersuchten Organismen) | UGA | Stop | Tryptophan |
| Mitochondrien von Säugern, Drosophila und S. cerevisiae und Protozoen | AUA | Isoleucin | Methionin = Start |
| Mitochondrien von Säugern | AGC, AGU | Serin | Stop |
| Mitochondrien von Säugern | AG(A, G) | Arginin | Stop |
| Mitochondrien von Drosophila | AGA | Arginin | Stop |
| Mitochondrien z. B. bei Saccharomyces cerevisiae | CU(U, C, A, G) | Leucin | Threonin |
| Mitochondrien Höherer Pflanzen | CGG | Arginin | Tryptophan |
| Einige Arten der Pilzgattung Candida | CUG | Leucin | Serin |
| Eukarya (selten) | CUG | Leucin | Start |
| Eukarya (selten) | ACG | Threonin | Start |
| Eukarya (selten) | GUG | Valin | Start |
| Bacteria | GUG | Valin | Start |
| Bacteria (selten) | UUG | Leucin | Start |
| Bacteria (SR1 Bacteria) | UGA | Stop | Glycin[23] |
Genetische Codes in DNA-Alphabet
[Bearbeiten | Quelltext bearbeiten]DNA-Sequenzdatenbanken wie GenBank geben auch mRNA-Sequenzen in einem historischen Konventionen entsprechenden Format an, bei dem das DNA-Alphabet verwendet wird, also T anstelle von U steht. Beispiele:[22]
- Standard Code (= id)
AS = FFLLSSSSYY**CC*WLLLLPPPPHHQQRRRRIIIMTTTTNNKKSSRRVVVVAAAADDEEGGGG Starts = ---M------**--*----M---------------M---------------------------- Base1 = TTTTTTTTTTTTTTTTCCCCCCCCCCCCCCCCAAAAAAAAAAAAAAAAGGGGGGGGGGGGGGGG Base2 = TTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGG Base3 = TCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAG
id = FFLLSSSSYY**CC*WLLLLPPPPHHQQRRRRIIIMTTTTNNKKSSRRVVVVAAAADDEEGGGG
- Vertebraten Mitochondrial Code
AS = FFLLSSSSYY**CCWWLLLLPPPPHHQQRRRRIIMMTTTTNNKKSS**VVVVAAAADDEEGGGG Starts = ----------**--------------------MMMM----------**---M------------ Base1 = TTTTTTTTTTTTTTTTCCCCCCCCCCCCCCCCAAAAAAAAAAAAAAAAGGGGGGGGGGGGGGGG Base2 = TTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGG Base3 = TCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAG
id = FFLLSSSSYY**CC*WLLLLPPPPHHQQRRRRIIIMTTTTNNKKSSRRVVVVAAAADDEEGGGG
- Hefe Mitochondrial Code
AS = FFLLSSSSYY**CCWWTTTTPPPPHHQQRRRRIIMMTTTTNNKKSSRRVVVVAAAADDEEGGGG Starts = ----------**----------------------MM---------------------------- Base1 = TTTTTTTTTTTTTTTTCCCCCCCCCCCCCCCCAAAAAAAAAAAAAAAAGGGGGGGGGGGGGGGG Base2 = TTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGG Base3 = TCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAG
id = FFLLSSSSYY**CC*WLLLLPPPPHHQQRRRRIIIMTTTTNNKKSSRRVVVVAAAADDEEGGGG
- Invertebraten Mitochondrial Code
AS = FFLLSSSSYY**CCWWLLLLPPPPHHQQRRRRIIMMTTTTNNKKSSSSVVVVAAAADDEEGGGG Starts = ---M------**--------------------MMMM---------------M------------ Base1 = TTTTTTTTTTTTTTTTCCCCCCCCCCCCCCCCAAAAAAAAAAAAAAAAGGGGGGGGGGGGGGGG Base2 = TTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGG Base3 = TCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAG
id = FFLLSSSSYY**CC*WLLLLPPPPHHQQRRRRIIIMTTTTNNKKSSRRVVVVAAAADDEEGGGG
AS = FFLLSSSSYY**CC*WLLLLPPPPHHQQRRRRIIIMTTTTNNKKSSRRVVVVAAAADDEEGGGG Starts = ---M------**--*----M------------MMMM---------------M------------ Base1 = TTTTTTTTTTTTTTTTCCCCCCCCCCCCCCCCAAAAAAAAAAAAAAAAGGGGGGGGGGGGGGGG Base2 = TTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGG Base3 = TCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAG
id = FFLLSSSSYY**CC*WLLLLPPPPHHQQRRRRIIIMTTTTNNKKSSRRVVVVAAAADDEEGGGG
Anmerkung: In der jeweils ersten Zeile „AS“ werden die Aminosäuren im Ein-Buchstaben-Code (siehe #Umgekehrte Codon-Tabelle) angegeben, wobei Abweichungen gegenüber dem Standard-Code (id) jeweils gefettet dargestellt sind (bzw. rot). In der zweiten Zeile „Starts“ zeigt M Initiation, * Termination; manche Varianten unterscheiden sich allein hinsichtlich (alternativer) Startcodons oder Stopcodons. Weitere Codes sind der frei zugänglichen Quelle zu entnehmen.[22]
Engineering des genetischen Codes
[Bearbeiten | Quelltext bearbeiten]Allgemein ist das Konzept von der Evolution des genetischen Codes vom ursprünglichen und mehrdeutigen genetischen Urcode zum wohldefinierten („eingefrorenen“) Code mit dem Repertoire von 20 (+2) kanonischen Aminosäuren akzeptiert.[24] Es gibt jedoch verschiedene Meinungen und Ideen, wie diese Änderungen stattfanden. Auf diesen basierend werden sogar Modelle vorgeschlagen, die „Eintrittspunkte“ für die Invasion des genetischen Codes mit synthetischen Aminosäuren voraussagen.[25]
Siehe auch
[Bearbeiten | Quelltext bearbeiten]Literatur
[Bearbeiten | Quelltext bearbeiten]- Lily E. Kay: Who wrote the book of life? A history of the genetic code. Stanford University Press, Stanford, Calif. 2000
- Deutsche Ausgabe: Das Buch des Lebens. Wer schrieb den genetischen Code? Aus dem amerikanischen Englisch übersetzt von Gustav Roßler. Suhrkamp, Frankfurt am Main 2005, ISBN 3-518-29346-X.
- Rüdiger Vaas: Der genetische Code. Evolution und selbstorganisierte Optimierung, Abweichungen und gezielte Veränderung. Wissenschaftliche Verlagsgesellschaft, Stuttgart 1994, ISBN 3-8047-1383-1.
- Lei Wang, Peter G. Schultz: Die Erweiterung des genetischen Codes. In: Angewandte Chemie. Band 117, Nr. 1, 2005, S. 34–68, doi:10.1002/ange.200460627.
Weblinks
[Bearbeiten | Quelltext bearbeiten]- Die Evolution des genetischen Codes: Neue Befunde untermauern das „stereochemische Modell“
- webmic.de
- Online DNA → Aminosäure Konverter
- Standard Code und Ausnahmen
- GeneCards: Human Gene Database (englisch)
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ a b c Johann Friedrich Miescher: Brief an Wilhelm His, 17. Dezember 1892 In: Miescher, Johann Friedrich: Die histochemischen und physiologischen Arbeiten, Band 1, Seite 116 f. Zitiert nach: Hans Blumenberg: Die Lesbarkeit der Welt.
- ↑ Albrecht Kossel: Harvey Lecture. Philadelphia 7, 33 (1911/1912). Hier angeführt nach: Carsten Bresch, Rudolf Hausmann: Klassische und molekulare Genetik. 3. Auflage. Berlin, Heidelberg, New York 1972. Springer Verlag. Seite 134. Bresch selbst nahm an, dass Albrecht Kossel den Schriftvergleich „erfunden“ habe.
- ↑ Hans Blumenberg: Die Lesbarkeit der Welt. Suhrkamp Verlag, Frankfurt am Main 1986. Kapitel XXII Der genetische Code und seine Leser. Seite 372 ff.
- ↑ Gino Segré, The big bang and the genetic code, Nature, Band 404, 2000, S. 437
- ↑ Gamow, Possible relation between DNA and protein structures, Nature, Band 173, 1954, S. 318
- ↑ Gamow, Possible mathematical relation between Deoxyribonucleic Acid and Proteins, Kong. Danske Vid. Selsk., Biolog. Meddelelser, Band 22, 1954, Nr. 3
- ↑ F. Crick, L. Barnett, S. Brenner, R. J. Watts-Tobin, General nature of the genetic code for proteins, Nature, Band 192, 1961, S. 1227–1232
- ↑ Judson, The eighth day of creation, Cold Spring Harbor Press 1996, S. 460f
- ↑ H. Drabkin, U. RajBhandary: Initiation of protein synthesis in mammalian cells with codons other than AUG and amino acids other than methionine. In: Molecular and Cellular Biology. Band 18, Nummer 9, September 1998, S. 5140–5147; PMID 9710598. PMC 109099 (freier Volltext).
- ↑ L. R. Cruz-Vera, M. A. Magos-Castro, E. Zamora-Romo, G. Guarneros: Ribosome stalling and peptidyl-tRNA drop-off during translational delay at AGA codons. In: Nucleic acids research. Band 32, Nummer 15, 2004, S. 4462–4468. doi:10.1093/nar/gkh784. PMID 15317870. PMC 516057 (freier Volltext).
- ↑ M. dos Reis, R. Savva, L. Wernisch: Solving the riddle of codon usage preferences: a test for translational selection. In: Nucleic acids research. Band 32, Nummer 17, 2004, S. 5036–5044. doi:10.1093/nar/gkh834. PMID 15448185. PMC 521650 (freier Volltext).
- ↑ U. Lagerkvist: "Two out of three": an alternative method for codon reading. In: Proceedings of the National Academy of Sciences. Band 75, Nummer 4, April 1978, S. 1759–1762. PMID 273907. PMC 392419 (freier Volltext).
- ↑ J. Lehmann, A. Libchaber: Degeneracy of the genetic code and stability of the base pair at the second position of the anticodon. In: RNA. Band 14, Nummer 7, Juli 2008, S. 1264–1269. doi:10.1261/rna.1029808. PMID 18495942. PMC 2441979 (freier Volltext).
- ↑ Markus Fricke, Ruman Gerst, Bashar Ibrahim, Michael Niepmann, Manja Marz: Global importance of RNA secondary structures in protein coding sequences. In: Bioinformatics. 7. August 2018, doi:10.1093/bioinformatics/bty678 (englisch).
- ↑ James Dewey Watson, Tania A. Baker, Stephen P. Bell, Alexander Gann, Michael Levine, Richard Losick, et al.: Molecular Biology of the Gene. 6. Auflage. Pearson/Benjamin Cummings, San Francisco 2008, ISBN 978-0-8053-9592-1, S. 521 ff.
- ↑ Erwin Schrödinger: What is life? The Physical Aspect of the Living Cell. 1944 (whatislife.stanford.edu [PDF] Based on lectures delivered under the auspices of the Dublin Institute for Advanced Studies at Trinity College, Dublin, in February 1943).
- ↑ Francis Crick: The Origin of the Genetic Code (= Journal of Molecular Biology. Band 38). Elsevier, 1968, ISSN 0022-2836, S. 367–79.
- ↑ Stefan Klein: Alles Zufall: Die Kraft, die unser Leben bestimmt. 2015, ISBN 978-3-499-61596-2 (books.google.de).
- ↑ C.R. Woese: On the Evolution of the Genetic Code. PNAS, 1965, S. 1546–1552, PMC 300511 (freier Volltext).
- ↑ Stephen J. Freeland, Laurence D. Hurst: Der raffinierte Code des Lebens. Spektrum der Wissenschaft, Juli 2004, S. 86–93.
- ↑ V. Kubyshkin, C. G. Acevedo-Rocha, N. Budisa: On universal coding events in protein biogenesis. In: Biosystems. 2017, doi:10.1016/j.biosystems.2017.10.004.
- ↑ a b c The Genetic Codes, laut NCBI letzte Aktualisierung: 18. November 2016; abgerufen am 25. Oktober 2017.
- ↑ J. H. Campbell, P. O’Donoghue u. a.: UGA is an additional glycine codon in uncultured SR1 bacteria from the human microbiota. In: Proceedings of the National Academy of Sciences. Band 110, Nummer 14, April 2013, S. 5540–5545. doi:10.1073/pnas.1303090110. PMID 23509275. PMC 3619370 (freier Volltext).
- ↑ Nediljko Budisa: The book at the Wiley Online Library. Wiley-VCH-Verlag, Weinheim 2005, ISBN 978-3-527-31243-6, doi:10.1002/3527607188 (englisch).
- ↑ V. Kubyshkin, N. Budisa: Synthetic alienation of microbial organisms by using genetic code engineering: Why and how? In: Biotechnologie Journal. 12. Jahrgang, 2017, S. 1600097, doi:10.1002/biot.201600097.
