Octopus Spring
| Octopus Spring | |
|---|---|

| |
| Lage | |
| Land oder Region | Teton County, Wyoming, USA |
| Koordinaten | 44° 32′ 2″ N, 110° 47′ 51″ W |
| Höhe | 2238 |
| Geologie | |
| Quelltyp | alkalische Thermalquelle, pH-Wert ≈ 8, Temperatur 90–95 °C |
| Austrittsart | Quelltopf |
| Hydrologie | |
| Flusssystem | Mississippi River |
| Vorfluter | White Creek → Firehole River → Madison River → Missouri River → Mississippi River → Golf von Mexiko |
| Schüttung | 75 – 100 ℓ/min |
Koordinaten: 44° 32′ 1,8″ N, 110° 47′ 50,7″ W
Octopus Spring (deutsch: Oktopus-Quelle) ist die Bezeichnung einer alkalischen heißen Quelle im unteren Geysir-Becken des Yellowstone-Nationalparks[1][2], knapp 10 km vom bekannten Geysir Old Faithful entfernt. Sie befindet sich im Teton County im Bundesstaat Wyoming der Vereinigten Staaten[3] auf einer Höhe von 2238 Meter über dem Meeresspiegel.[A. 1]
Die Quelle hat ihren Namen von den strahlenförmig wie die Arme eines Oktopus verlaufenden Abflusskanälen.[1]
Die aus Quellen im Yellowstone-Nationalpark isolierten Bakterien und Archaeen sind aufgrund ihres an die hohen Umgebungstemperaturen angepassten Stoffwechsels von Interesse für die Biochemie. So vertreibt der Schweizer Konzern Roche beispielsweise die in Bakterien der Spezies Thermus aquaticus vorkommende thermostabile DNA-Polymerase[5] – diese Bakterien wurden zuerst im benachbarten Mushroom Spring gefunden, später auch hier. Er erzielt damit Einnahmen in Milliardenhöhe;[6] denn sie findet Verwendung bei der Polymerase-Kettenreaktion (PCR) der DNA. Nach einem Gerichtsurteil aus dem Jahre 2000 ist der National Park Service ermächtigt, künftig an Forschungsergebnissen, die im Yellowstone-Nationalpark erzielt wurden, finanziell teilzuhaben.[7][8]
Geographie
[Bearbeiten | Quelltext bearbeiten]Die Quelle entwässert über den White Creek in den Firehole River.[2] Im Gebiet rund um die Quelle (dem unteren Geysir-Becken) ist es überwiegend eben und es gibt noch etliche andere Quellen[A. 2] wie etwa den Great-Fountain-Geysir, den Firehole Spring, den Surprise Pool[2] sowie den Mushroom Spring.[9] Das Quellgebiet liegt in einem Tal, um dieses Gebiet herum ist das Gelände größtenteils hügelig,[A. 3] der höchste Punkt in der Umgebung hat eine Höhe von 2368 Metern und liegt etwa 1,1 km im Osten.[A. 2]
Die Besiedlungsdichte um die Quelle ist sehr gering, d. h., es leben dort weniger als 2 Menschen pro Quadratkilometer;[10] es gibt weder eine Stadt noch auch nur eine Ortschaft in der Nähe.[A. 2][11][2]
Geologie
[Bearbeiten | Quelltext bearbeiten]
Der Octopus Spring wird durch die vom Magma unter der Oberfläche erzeugte Hitze erwärmt. Das Quellwasser und die darin gelösten Stoffe haben ihren Ursprung in einer tiefen hydrothermalen Quelle (mit ca. 195 °C in ~150 m Tiefe). Sie stellt damit eine der Austrittsöffnungen für organische und anorganische Komponenten im gesamten hydrothermalen System dar.[12] Das heiße Wasser löst das Siliziumdioxid (SiO2) des Vulkangesteins auf und fällt es aus, wodurch die Silikatablagerungen der Sinter eine grauweiße Farbe erhalten.[13][14] Die Quelle selbst hat eine Temperatur von 90–95 °C (Grad Celsius),[14][1][12] in den Abflusskanälen kühlt sich das Wasser auf 83 °C ab,[1] in der Nähe der Quelle dann bis auf ca. 60 °C.[14]
Das Wasser ist arm an Schwefelwasserstoff (H2S) und alkalisch mit einem pH-Wert von etwa 8.[14]
Klima
[Bearbeiten | Quelltext bearbeiten]Das Klima ist kalt gemäßigt (boreal),[15] die Durchschnittstemperatur der Luft beträgt −1 °C. Der wärmste Monat ist der Juli mit 16 °C und der kälteste der Dezember mit −15 °C.[16] Die durchschnittliche Niederschlagsmenge beträgt 401 Millimeter pro Jahr. Der nasseste Monat ist der Mai mit 51 Millimeter Regen, der trockenste der Juni mit 23 Millimetern.[17]
Ökologie
[Bearbeiten | Quelltext bearbeiten]Octopus Spring ist fast vollständig von Kiefernwald umgeben.[11]
Der gleichmäßige Abfluss von schätzungsweise 75 bis 100 Litern pro Minute ernährt ausgedehnte und farbenfrohe mikrobielle Matten.[1]
Etwa 2 Meter vom kristallblauen Becken entfernt erscheinen in den Abflusskanälen rosafarbene, filamentöse mikrobielle Lebensgemeinschaften (Konsortien). Weiter unten in den Kanälen sinken die Temperaturen, und die mikrobiellen Matten-Konsortien verändern sich sowohl in ihrer Farbe als auch in ihrer Zusammensetzung.[1]
Insgesamt bestehen die Matten des Octopus Spring aus einer Vielzahl verschiedener Mikroben, darunter photoautotrophe (Energie durch Photosynthese gewinnend), chemoautotrophe (anorganische Stoffe, wie z. B. Wasserstoff nutzend) und heterotrophe (wie die meisten Tiere organische Stoffe benötigende) Organismen. Der Octopus Spring weist eine hohe Konzentration an Wasserstoffgas (H2) auf, das von spezialisierten Bakterien verwertet wird.[14][18] Zu den Bakterien, die an den mikrobiellen Lebensgemeinschaften beteiligt sind, gehören außerdem Cyanobakterien, Grüne Schwefelbakterien (Chlorobien) und Grüne Nicht-Schwefelbakterien (Chloroflexi).[1][14][18]
Die zellulären Mikroorganismen des Octopus Spring – bekannt sind nur Prokaryoten (Bakterien und Archaeen) – werden zudem von Viren parasitiert,[19] deren Erforschung allerdings noch am Anfang steht[19] (Stand März 2023).
Beispiele:
Cyanobakterien
[Bearbeiten | Quelltext bearbeiten]Synechococcus (historisch)
[Bearbeiten | Quelltext bearbeiten]Synechococcus ist eine Gattung von Cyanobakterium der Familie Synechococcaceae und Ordnung Synechococcales. Eine Studie aus dem Jahr 2006 von Anne-Soisig Steunou et al. unterschied vier verschiedene Ökotypen dieser Gattung (A bis D) sowie weitere Untertypen derselben. Zwei dieser Typen (A und B') waren als thermophile Synechococcus-Varianten aus dem Octopus Spring (daher auch als OS-A und OS-B' bezeichnet). Auch eine weitere Studie aus demselben Jahr von Jessica Allewalt et al. untersuchte die hier als Genotypen bezeichneten varianten A und B des Octopus Spring (mit Untervarianten).
Diese beiden Typen wiesen von allen in diesem Ökosystem vorhandenen Cyanobakterien die höchste Temperaturtoleranz auf, nämlich bis zu etwas über 70 °C. Dabei waren A-ähnliche Sequenzen bei etwas höheren Temperaturen und B-ähnliche Sequenzen bei etwas niedrigeren Temperaturen verbreitet.[18][20][14] Diese Mikroorganismen zeigten sich zur Stickstofffixierung fähig, dabei haben sie diese und die Photosynthese zeitlich voneinander getrennt – beide Prozesse sind nämlich nicht miteinander verträglich und können nicht zur selben Zeit am selben Ort ablaufen. Um das zu bewerkstelligen, besitzen die betrachteten Cyanobakterien entscheidende regulatorische Schalter, die diese beiden und andere Stoffwechselprozesse innerhalb der Zellen steuern. Insbesondere verfügen sie wie Knöllchenbakterien über nif-Gene (nif ist ein Akronym aus lateinisch Nitrogenium ‚Stickstoff‘, N2; und Fixierung). Diese Gene werden exprimiert, wenn die Matte bei abnehmender Helligkeit am späten Nachmittag anoxisch wird. Damit einher geht eine Zunahme der Nitrogenaseaktivität, die während der Nacht N2 bindet.[18][14][20] Die Fixierung von N2 erfordert viel Energie. Um diese Energie in der Nacht zu gewinnen, sind die Bakterien auf Fermentationsprozesse (Gärung) angewiesen.[18][14][20]
Tagsüber setzen die thermophilen Synechococcus-Typen auf Photosynthese und Zellatmung, sodass der Gehalt an Sauerstoff (O2) hoch ist; nur nachts schalten sie auf Fermentation und N2-Fixierung um.[14][20]
Thermostichus bzw. „Synechococcus_A“
[Bearbeiten | Quelltext bearbeiten]Im Jahr 2020 schlugen Jiří Komárek et al. eine Reorganisation der damals sehr umfangreichen Gattung Synechococcus vor, da die ihr damals zugerechneten Spezies und Stämme untereinander durch starke Abweichungen in der 16S rRNA gekennzeichnet waren und eine Ähnlichkeit nur in ihrer äußeren Gestalt bestand. Im Zug dieser Reorganisation wurden insbesondere die thermophilen Varianten (Spezies, aber auch einige einzelne Stämme) abgetrennt und in die neuen Gattungen Thermosynechococcus und Thermostichus verschoben, inklusive einer neuen Familien- und Ordnungszuweisung.[21] Einzelheiten dieser Reorganisation sind noch in der Diskussion: Während die Taxonomie des National Center for Biotechnology Information genauso wie die LPSN die Gattung Thermostichus in einer weiteren eigenen Familie Thermostichaceae und Ordnung Thermostichales sieht, ordnet die Genome Taxonomy Database (GTDB) die thermophilen ehemaligen Synechococcus-Mitglieder derselben Familie und Ordnung zu wie Thermosynechococcus.[A. 4]
Insbesondere gilt dies für die früher als Synechococcus lividus bezeichnete thermophile Spezies. Ihr Referenzstamm PCC 6175T wurde auf der Promontory-Halbinsel im Süden des Yellowstone Lake gefunden,[25][26][27] der ihr ebenfalls zugehörige Stamm C1 aber im Octopus Spring.[28] Diese Spezies wird mit diesen beiden Stämmen in der NCBI-Taxonomie heute als Thermostichus lividus in die Gattung Thermostichus der Familie Thermostichaceae, Ordnung Thermostichales gestellt.[25][28][A. 5]
In der GTDB wird die den Stamm PCC 6715 enthaltende Gattung mit dem provisorischen Namen „Synechococcus_A“ bezeichnet, die Spezies selbst als „Synechococcus_A lividus“ (Referenzstamm nach wie vor PCC 6715).[29] Als enthaltende Familie gilt dort jedoch die Thermosynechococcaceae (mit Typusgattung Thermosynechococcus[24][22]) und als Ordnung Thermosynechococcales.[23] Klasse und Phylum sind in beiden Taxonomien dieselben wie bei Synechococcus, wenn auch mit unterschiedlichen Bezeichnungen und Umfang (Cyanophyceae syn. Cyanobacteria respektive Cyanobacteria syn. Cyanobacteriota, siehe Artikel Cyanobakterien).
Grüne Nichtschwefelbakterien
[Bearbeiten | Quelltext bearbeiten]Unter den Grünen Nichtschwefelbakterien (Choroflexi) fanden sich hier (wie auch im Mushroom Spring) Vertreter der Gattungen Roseiflexus, Chloroflexus sowie Anaerolineae-ähnliche Arten.[30]
Grüne Schwefelbakterien
[Bearbeiten | Quelltext bearbeiten]Unter den Grünen Schwefelbakterien (Chlorobi) fanden sich hier (ebenfalls wie auch im Mushroom Spring) „Candidatus Thermochlorobacter aerophilus“[31] syn. „Ca. T. aerophilum“,[31][32][33] Familie Thermochlorobacteraceae bzw. Chloroherpetonaceae in der Ordnung Chlorobiales. Referenzstamm ist OS GSB[31] (oder kurz OS[32][33]).[30]
Deinococcus-Thermus
[Bearbeiten | Quelltext bearbeiten]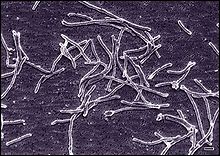
1995 berichteten Stephen Nold und David Ward über die Identifizierung von Bakterien der Gattung Thermus im Octopus Spring, darunter die Spezies Thermus aquaticus und Thermus ruber[34] (sowie zwei weiteren);[35] und konnten damit frühere Hinweise von um 1985 bestätigten.[36][37] Bakterien dieser Gattung aus dem Phylum Deinococcus-Thermus, wie der bereits in den späten 1960er Jahren in der nahe gelegenen Mushroom Spring gefundene Stamm Thermus aquaticus YT-1,[38][39] sind von besonderer Bedeutung. Sie besitzen eine thermostabile DNA-Polymerase (wie hier die Taq-Polymerase), mit deren Hilfe erstmals 1983 Kary Mullis die PCR (polymerase chain reaction) so weit verbesserte und weiterentwickelte, dass sie in großem Umfang in der Praxis eingesetzt werden konnte.[40][41][38]
Aquificaceae
[Bearbeiten | Quelltext bearbeiten]Thermocrinis ruber ist ein gramnegatives Bakterium aus der Familie Aquificaceae, das die Form eines Stäbchens hat und durch Rotation seiner polytrich-monopolaren Geißeln beweglich ist. Der Referenzstamm OC 1/4 (alias DSM 12173) stammt vom Octopus Spring.[42] Die Zellen von T. ruber wachsen vorwiegend als lange Fäden auf festen Oberflächen. Das Bakterium kann in einem Temperaturbereich zwischen 44 °C und 89 °C überleben, wobei die optimale Temperatur bei 80 °C liegt. Mit einem hohen GC-Gehalt von etwa 47 % können sich die Bakterien gut an eine Umgebung mit hohen Temperaturen anpassen. Das Bakterium kann eine Vielzahl von Nährstoffquellen nutzen, darunter molekularen Wasserstoff (H2), Thiosulfat, elementaren Schwefel, Formiat und Formamid als Elektronendonatoren, Sauerstoff als Elektronenakzeptor und Kohlendioxid (CO2), Formiat und Formamid als Kohlenstoffquellen.[43][14] Die Fähigkeit der Bakterien, viele Ressourcen als Nahrung und Energie zu nutzen, ermöglicht es ihnen, in der extremen Umgebung dieser Quelle zu überleben.[14]
Bacillota
[Bearbeiten | Quelltext bearbeiten]Thermoanaerobacter acetoethylicus (früher Thermobacteroides acetoethylicus), ist eine Spezies thermophiler, nicht sporen bildender Bakterien[44][45] aus dem Phylum Bacillota (alias Firmicutes), die hier in der Thermalquelle entdeckt und erstmals isoliert wurde. Diese Bakterien produzieren Ethanol und Essigsäure als Fermentationsprodukte, aber keine Milchsäure.[45] Der Wachstumsbereich von T. acetoethylicus liegt bei 40–80 °C und einem pH-Wert von 5,5–8,5, wobei die optimale Wachstumstemperatur bei etwa 65 °C liegt.[44] Die Art wurde 1981 ursprünglich in eine eigene neue Gattung Thermobacteroides eingeordnet; auf Grundlage weiterer Studien dann aber 1993 in die Gattung Thermoanaerobacter verschoben.[45]
Acidobacteriota
[Bearbeiten | Quelltext bearbeiten]Entdeckt wurde zudem „Candidatus Chloracidobacterium thermophilum“, diese Kandidatenspezies wird heute meist den Blastocatellia (früher genannt Acidobacteria subdivision 4)[46][47] (oder den Terriglobia[48]) in Phylum Acidobacteriota zugerechnet.[46][47][48][30] Referenzstamm ist B alias ATCC BAA-2647 oder JCM 3019.[48]
Candidatus Calescibacteriota
[Bearbeiten | Quelltext bearbeiten]Aus der Metagenomik bzw. per Einzelzell-Sequenzierung stammen Hinweise, dass in der Quelle auch Vertreter des Kandidatenphylums Calescibacteriota (auch Calescibacterota oder Calescamantes genannt) vorhanden sind. Eine frühere Bezeichnung dieser Gruppe ist Candidate division EM19 (auch EM 19 geschrieben) nach dem hier gefundenen Vertreter Bacterium EM-19 (GenBank U05662.1).[49][50] Ein weiterer dieser Vertreter ist Calescamantes bacterium T3.1,[51] aus dem Sediment stammt ein als IMG:3300001339 bezeichnetes MAG (Metagenom).[52]
Candidatus Hydrothermae
[Bearbeiten | Quelltext bearbeiten]Zu dem als Hydrothermae bezeichneten Kandidatenphylum (provisorische Bezeichnung Candidate division EM3, auch EM 3)[53] gehört das Pink filament bacterium EM-3.[49][54]
Methanothermobacter
[Bearbeiten | Quelltext bearbeiten]Zu den in der Quelle gefundenen Archaeen gehört Methanothermobacter thermautotrophicus (früher als Methanobacterium thermautotrophicum der Gattung Methanobacterium zugerechnet). Dies ist ein streng anaerobes autotrophes extremophiles Archaeon, das die Form unregelmäßig gebogener Stäbchen hat und oft auch lange Fäden bildet.[55][14] M. thermautotrophicus hat ein Temperaturoptimum für Wachstum und Methanogenese von 65 °C bis 70 °C. Die Organismen nutzen Ammoniak als Stickstoffquelle, Sulfid als Schwefelquelle, Wasserstoff als Hauptenergiequelle und Kohlendioxid als Kohlenstoffquelle. Ihre Fähigkeit, Wasserstoff als Reduktionsmittel sowie Kohlendioxid zu nutzen, klassifiziert sie als wasserstoff-oxidierende Autotrophe. Die Fähigkeit ihrer Ribosomen, ihre Hyperchromizität um 20 % zu erhöhen und große Hitze zu ertragen, spielt eine wichtige Rolle für die maximale Wachstumstemperatur in Abhängigkeit von der Umgebung.[55][14] Insgesamt scheint sich M. thermautotrophicus optimal an die Umgebungstemperaturen anzupassen.[56][14]
Sulfolobus
[Bearbeiten | Quelltext bearbeiten]
Es gibt darüber hinaus auch Hinweise auf Archaeen aus der Tack-Supergruppe im Octopus Spring: „Sulfolobus sp. (from Octopus Spring 1)“.[57][36] Ob auch im Octopus Spring vorhandene Sulfolobus-Arten dort von Sulfolobus-Virus STSV1 parasitiert werden, ist nicht bekannt; es wäre aber möglich, dass dieses oder verwandte Archaeenviren vorkommen.
„Pyrovirus“
[Bearbeiten | Quelltext bearbeiten]Zwar kommen Aquificae-Bakterien (wie Thermocrinis) in vielen Geothermal-Systemen vor, aber sie sind nur schwer zu kultivieren. Das ist die Ursache, dass das erste Aquificae-Virus erst im Jahr 2020 beschrieben wurde. In diesem Jahr identifizierten Marike Palmer, Simon Roux und Kollegen per Metagenomik das von ihnen als OS3173 bezeichnete Virus-Isolat mit einem zirkulären dsDNA-Genom. Für dieses Virus schlugen die Autoren die vorläufige Artbezeichnung „Thermocrinis Octopus Spring virus“ (TOSV, nicht zu verwechseln mit Toskana-Virus) vor. Sie stellten diesen Fund zusammen mit acht verwandten unkultivierten Virus-Genomen (UViGs) aus ähnlichen Umgebungen, die drei weitere Spezies repräsentieren in eine gemeinsame Virusgattung mit vorgeschlagener Bezeichnung „Pyrovirus“ innerhalb der Klasse Caudoviricetes (Caudoviren, damals noch als Ordnung Caudovirales angesehen). Dies begründeten die Autoren damit, dass trotz der geringen Gesamtähnlichkeit der Viren aus verschiedenen heißen Quellen ihre Genome einen hohen Grad an Syntenie zeigten und zahlreiche Gene für den Nukleotidstoffwechsel gemeinsam hatten, darunter ein DNA-Polymerase-Polyprotein vom PolA-Typ, eine DNA-Gyrase, eine Helikase, eine DNA-Methylase und weitere. Außerdem konnten konservierte Gene ausgemacht werden, u. a. für das Viruskapsid, was auf eine entfernte Verwandtschaft mit kultivierten Caudoviren hindeutete. Weiter Gründe für die vorgeschlagene Taxonomie waren die vorhergesagte atypische bidirektionalen Replikationsstrategie und die geringe Sequenzähnlichkeit mit bekannten viralen Genomen.[19]
Da mehrere CRISPR-Spacer, die mit den viralen Genomen übereinstimmen, in den Genomen und metagenomischen Contigs der Aquificae-Bakterien identifiziert wurden, nimmt man an, dass diese Viren Aquificae infizieren.[19]
Die Ergebnisse wurden durch weitere, 2021 und 2022 veröffentlichte Arbeiten bestätigt.[58][59]
Anmerkungen
[Bearbeiten | Quelltext bearbeiten]- ↑ Berechnet aus Höhendaten (DEM 3") mit Viewfinder Panoramas.[4] Der vollständige Algorithmus ist hier verfügbar.
- ↑ a b c In einer Umgebung von 20 Kilometern im Vergleich zur durchschnittlichen Bevölkerungsdichte laut GeoNames.[3]
- ↑ Berechnet aus dem Schnittpunkt aller Höhendaten (DEM 3") mit Viewfinder Panoramas, im Umkreis von 10 Kilometern.[4] Der vollständige Algorithmus ist hier verfügbar.
- ↑ Die Gattung Thermosynechococcus wird jetzt gewöhnlich der neuen Familie Thermosynechococcaceae und Ordnung Thermosynechococcales als Typusgattung zugerechnet.[22][23] (Mit Ausnahme in der Prokaryotic names with Standing in Nomenclature[24]).
- ↑ Lediglich die beiden früher Synechococcus lividus zugerechneten Stämme PCC 6176 und PCC 6717 verblieben ohne Artzuweisung bei der Gattung Synechococcus.
Weblinks
[Bearbeiten | Quelltext bearbeiten]- Sarah Bordenstein: Octopus Spring. Microbial Life, Science Education Resource Center, Carleton College. Stand: 14. März 2023.
- Mark C. Capece, Evan Clark, Jamal K. Saleh, Daniel T. Halford, Nicole Heinl, Samuel Hoskins, Lynn Rothschild: Polyextremophiles and the Constraints for Terrestrial Habitability. In: Polyextremophiles, April 2013, S. 3–59; doi:10.1007/978-94-007-6488-0_1, ResearchGate. Siehe insbes. Fig. 6: Octopus Spring.
- Thomas D. Brock: The Road to Yellowstone – And Beyond. In: Annual Reviews Microbiol., Band 49, 1995, S. 1–28; doi:10.1146/annurev.mi.49.100195.000245.
- Anna-Louise Reysenbach, Gene S. Wickham, Norman R. Pace: Phylogenetic analysis of the hyperthermophilic pink filament community in Octopus Spring, Yellowstone National Park. In: ASM Journals: Applied and Environmental Microbiology, Band 60, Nr. 6, 1. Juni 1994, S. 2113-2119; doi:10.1128/aem.60.6.2113-2119.1994, SERC(Carleton College):13149 (englisch).
- Allan Treiman: Extremities: Geology and Life in Yellowstone and Implications for Other Worlds. A Fieldtrip and Workshop for 6th–12th Grade Teachers – hosted by David Ward. Juli 26 – August 2, 2002. Extremities Workshop: Index. – Stand: 15. November 2002.
…
Octopus Springs:- Octopus Spring – 1 – Hot Water – Stand: 15. November 2002.
- Octopus Spring - 2 – Cool Water – Stand: 15. November 2002.
- Microbiology Laboratory – 2 – mit: Aquifex, Thermotoga, Synechococcus – Stand: 15. November 2002.
- Microbiology Laboratory – 3 – mit: Chloroflexus – Stand: 15. November 2002.
- …
Universities Space Research Association (USRA): Lunar and Planetary Institute (LPI)
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ a b c d e f g Sarah Bordenstein: Octopus Spring. Auf: Microbial Life, Stand: 2. März 2023.
- ↑ a b c d Octopus Spring. Auf Mapcarta (de).
- ↑ a b Octopus Spring. Auf Geonames.org (cc-by)
- ↑ a b Viewfinder Panoramas Digital elevation Model. 21. Juni 2015 (englisch).
- ↑ Roche: PCR – eine ausgezeichnete Methode ( vom 27. Januar 2007 im Internet Archive) (pdf – 28. Oktober 2007; 353 kB),
- ↑ Park Service may license microbe use. Auf: Billings Gazette vom 5. Oktober 2008. Memento im Webarchiv vom 23. März 2023.
- ↑ National Park Service: Benefit-Sharing in the National Parks Environmental Impact Statement (28. Oktober 2007). Memento im Webarchiv vom 10. Februar 2005.
- ↑ National Park Service: General Conditions for Scientific Research and Collecting Permit (28. Oktober 2007). Memento im Webarchiv vom 23. März 2023.
- ↑ Vera Thiel1, Michael Hügler, David M. Ward, Donald A. Bryant: The Dark Side of the Mushroom Spring Microbial Mat: Life in the Shadow of Chlorophototrophs. II. Metabolic Functions of Abundant Community Members Predicted from Metagenomic Analyses. In: Frontiers in Microbiology, Band 8, 6. Juni 2017; doi:10.3389/fmicb.2017.00943.
- ↑ NASA Earth Observations: Population Density. NASA/SEDAC, archiviert vom am 9. Februar 2016 (englisch). Memento im Webarchiv vom 9. Februar 2016.
- ↑ a b NASA Earth Observations: Land Cover Classification. NASA/MODIS, archiviert vom am 28. Februar 2016 (englisch). Memento im Webarchiv vom 28. Februar 2016.
- ↑ a b Michael Gonsior, Norbert Hertkorn, Nancy Hinman, Sabine E.-M. Dvorski, Mourad Harir, William J. Cooper, Philippe Schmitt-Kopplin: Yellowstone Hot Springs are Organic Chemodiversity Hot Spots. In: Nature: Scientific Reports, Band 8, Nr. 14155, 20. September 2018; doi:10.1038/s41598-018-32593-x, PDF.
- ↑ Joyce Cheatham, Erica L. Glover, Lollie Garay, Mark McGaugh, Rob Shauger: Octopus Springs. Yellowstone National Park. Lunar and Planetary Institute. 2007. Memento im Webarchiv vom 4. Dezember 2008.
- ↑ a b c d e f g h i j k l m n o Yellowstone Hot Springs §Yellowstone Hot Spring Regions. Auf MicrobeWiki, Kenyon College, Department of Biology. Stand: 20. August 2010. Anmerkung: Klassifizierung und Bezeichnungsweise wurden im Artikel hier aktualisiert/korrigiert.
- ↑ M. C. Peel, B. L. Finlayson: Updated world map of the Köppen-Geiger climate classification. In: Hydrology and Earth System Sciences. 11. Jahrgang, Nr. 5. EGU, 2007, S. 1633–1644, doi:10.5194/hess-11-1633-2007 (englisch).
- ↑ NASA Earth Observations Data Set Index. NASA, archiviert vom am 3. Februar 2016 (englisch). Memento im Webarchiv vom 3. Februar 2016.
- ↑ NASA Earth Observations: Rainfall (1 month - TRMM). NASA/Tropical Rainfall Monitoring Mission, archiviert vom am 12. April 2016 (englisch). Memento im Webarchiv vom 12. April 2016.
- ↑ a b c d e
Anne-Soisig Steunou, Devaki Bhaya, Mary M. Bateson, Melanie C. Melendrez, David M. Ward, Eric Brecht, John W. Peters, Michael Kühl, Arthur R. Grossman: In situ analysis of nitrogen fixation and metabolic switching in unicellular thermophilic cyanobacteria inhabiting hot spring microbial mats. In: PNAS, Band 103, Nr. 7, 7. Februar 2006, S. 2398-2403; doi:10.1073/pnas.0507513103, PMID 16467157, PMC 1413695 (freier Volltext). Dazu:
- Arthur Grossman: Hot-Spring Bacteria Flip a Metabolic Switch mit Abb.: Octopus Spring. Auf: EurekAlert! vom 30. Januar 2006. Die Abb. ist ein Memento im Webarchiv vom 7. Mai 2006.
- ↑ a b c d Marike Palmer, Brian P. Hedlund, Simon Roux, Philippos K. Tsourkas, Ryan K. Doss, Casey Stamereilers, Astha Mehta, Jeremy A. Dodsworth, Michael Lodes, Scott Monsma, Tijana Glavina del Rio, Thomas W. Schoenfeld, Emiley A. Eloe-Fadrosh, David A. Mead: Diversity and Distribution of a Novel Genus of Hyperthermophilic Aquificae Viruses Encoding a Proof-Reading Family-A DNA Polymerase. In: Frontiers in Microbiology, Band 11, Nr. 583361, 12. November 2020; doi:10.3389/fmicb.2020.583361, PMC 7689252 (freier Volltext), PMID 33281778.
- ↑ a b c d Jessica P. Allewalt, Mary M. Bateson, Niels Peter Revsbech, Kimberly Slack, David M. Ward: Effect of Temperature and Light on Growth of and Photosynthesis by Synechococcus Isolates Typical of Those Predominating in the Octopus Spring Microbial Mat Community of Yellowstone National Park. In: ASM Journals: Applied and Environmental Microbiology, Band 72, Nr. 1, 1. Januar 2006; doi:10.1128/AEM.72.1.544-550.2006, PMID 16391090, PMC 1352173 (freier Volltext).
- ↑ Jiří Komárek, Jeffrey R. Johansen, Jan Šmarda, Otakar Strunecký: Phylogeny and taxonomy of Synechococcus-like cyanobacteria. In: Fottea, Band 20, Nr. 2, S. 171–191; doi:10.5507/fot.2020.006.
- ↑ a b NCBI Taxonomy Browser: Thermosynechococcus.
- ↑ a b GTDB: Thermosynechococcus (genus).
- ↑ a b LPSN: Genus "Thermosynechococcus" Katoh et al. 2001.
- ↑ a b NCBI Taxonomy Browser: Thermostichus lividus PCC 6715 (strain), syn. Synechococcus lividus PCC 6715. Dazu: NCBI Nucleotide: Thermostichus lividus PCC 6715 chromosome, …. Die angegebenen Koordinaten zeigen auf die Promontory-Halbinsel.
- ↑ Promontory Tip. Auf Mapcarta (de).
- ↑ The Promontory. Auf: Google Maps.
- ↑ a b NCBI Taxonomy Browser: Thermostichus lividus C1 (strain), syn. Synechococcus lividus C1. Dazu: NCBI Nucleotide: Synechococcus lividus C1 isolation-source Octopus Spring cyanobacterial mat, Yellowstone National Park ….
- ↑ GTDB: Synechococcus lividus (species). Dazu: GCF_002754935.1: Synechococcus_A lividus (NCBI strain id: PCC 6715).
- ↑ a b c Zhenfeng Liu, Christian G. Klatt, Marcus Ludwig, Douglas B. Rusch, Sheila I. Jensen, Michael Kühl, David M. Ward, Donald A. Bryant: ‘Candidatus Thermochlorobacter aerophilum:’ an aerobic chlorophotoheterotrophic member of the phylum Chlorobi defined by metagenomics and metatranscriptomics. In: Nature: The ISME Journal, Band 6, S. 1869–1882, 29. März 2012; doi:10.1038/ismej.2012.24, PMID 22456447, PMC 3446795 (freier Volltext).
- ↑ a b c LPSN: Species "Candidatus Thermochlorobacter aerophilus", synonym "Candidatus Thermochlorobacter aerophilum" – Type strain: OS GSB.
- ↑ a b GTDB: Thermochlorobacter aerophilum (species), dazu: GCA_002794105.1: Thermochlorobacter aerophilum – NCBI strain identifiers: OS.
- ↑ a b NCBI Taxonomy Browser: Candidatus Thermochlorobacter aerophilum (species), dazu: NCBI Nucleotide: MAG: Candidatus Thermochlorobacter aerophilum isolate OS,…. Isolation source: Yellowstone National Park Mushroom and Octopus hot spring.
- ↑ LPSN: Species "Thermus flavus"
- ↑ Stephen C. Nold, David M. Ward: Diverse Thermus Species Inhabit a Single Hot Spring Microbial Mat. In: Systematic and Applied Microbiology, Band 18, Nr. 2, 1. Januar 1995, S. 274–278; doi:10.1016/s0723-2020(11)80398-x, PMID 11539573. Anm.: Abhängig von der Plattform kann im Titel Thermus als Thermlus verschrieben sein (korrekt auf PubMed und im PDF).
- ↑ a b D. A. Stahl, D. J. Lane, G. J, Olsen, N. R. Pace: Characterization of a Yellowstone hot spring microbial community by 5S rRNA sequences. In: ASM Journals: Applied and Environmental Microbiology, Band 49, Nr. 6, 1. Juni 1985; doi:10.1128/aem.49.6.1379-1384.1, PMID 2409920, PMC 241732 (freier Volltext).
- ↑ NCBI Nucleotide:
- ↑ a b Catherine C. Helm-Clark: MERRY BLOGMAS FOR DECEMBER 9: DNA AND YELLOWSTONE. THE BIRTHPLACE OF DNA FINGERPRINTING BY THE PCR METHOD. Blog auf: Gnarly Science vom 9. Dezember 2017.
- ↑ Thomas D. Brock, Hudson Freeze: Thermus aquaticus gen. n. and sp. n., a nonsporulating extreme thermophile. In: ASM Journals: Journal of Bacteriology, Band 98, Nr. 1, S. 289–297, April/August 1969; doi:10.1128/jb.98.1.289-297.1969, PMID 5781580, PMC 249935 (freier Volltext), PDF. Siehe insbes. Tbl. 1 (Fundort YT-1: Mushroom Spring, Station I).
- ↑ Thermus. Lexikon der Biologie auf spektrum.de, 1999.
- ↑ Taq-DNA-Polymerase. Lexikon der Biologie auf spektrum.de, 1999.
- ↑ Thermocrinis albus OC 1/4. Auf: Bacdive.
- ↑ Robert Huber, Wolfgang Eder, Stefan Heldwein, Gerhard Wanner, Harald Huber, Reinhard Rachel, Karl O. Stetter: Thermocrinis ruber gen. nov., sp. nov., a Pink-Filament-Forming Hyperthermophilic Bacterium Isolated from Yellowstone National Park. In: Applied and Environmental Microbiology (AEM), Band 65, Nr. 10, S. 3576–3583, 1. Oktober 1998; doi:10.1128/AEM.64.10.3576-3583.1998, PMID 9758770, PMC 106467 (freier Volltext).
- ↑ a b Arie Ben-Bassat, Joseph G. Zeikus: Thermobacteroides acetoethylicus gen. nov. and spec. nov., a new chemoorganotrophic, anaerobic, thermophilic bacterium. In: Archives of Microbiology, Band 128, Nr. 4, 1981, S. 365–370; doi:10.1007/BF00405914.
- ↑ a b c Fred A. Rainey, Erko Stackebrandt: Transfer of the Type Species of the Genus Thermobacteroides to the Genus Thermoanaerobacter as Thermoanaerobacter acetoethylicus (Ben-Bassat and Zeikus 1981) comb. nov., Description of Coprothermobacter gen. nov., and Reclassification of Thermobacteroides proteolyticus as Coprothermobacter proteolyticus (Ollivier et al. 1985) comb. nov. In: International Journal of Systematic Bacteriology, Band 43, Nr. 4, Oktober 1993, S. 857–859; doi:10.1099/00207713-43-4-857.
- ↑ a b NCBI Taxonomy Browser: Chloracidobacterium thermophilum (species), dazu: NCBI Nucleotide: Chloracidobacterium thermophilum strain B … USA: Octopus Spring, Yellowstone National Park, Wyoming.
- ↑ a b GTDB: Chloracidobacterium thermophilum (species).
- ↑ a b c LPSN: Species "Chloracidobacterium thermophilum" – Type strain: ATCC BAA-2647; B; JCM 30199.
- ↑ a b Anna-Louise Reysenbach, Gene S. Wickham, Norman R. Pace: Phylogenetic analysis of the hyperthermophilic pink filament community in Octopus Spring, Yellowstone National Park. In: ASM Journals: Applied and Environmental Microbiology, Band 60, Nr. 6, 1. Juni 1994, S. 2113-2119; doi:10.1128/aem.60.6.2113-2119.1994, PMID 7518219, PMC 201609 (freier Volltext), SERC(Carleton College):13149 (englisch).
- ↑ NCBI Nucleotide: Eubacteria EM 19 16S ribosomal RNA, partial sequence, GenBank: U05662.1.
- ↑ NCBI Taxonomy Browser: Calescamantes bacterium T3.1 (species). Dazu: Nucleotide: Calescamantes bacterium T3.1.
- ↑ Eric Daniel Becraft, Jeremy A. Dodsworth, Senthil K. Murugapiran, J. Ingemar Ohlsson, Brandon R. Briggs, Jad Kanbar, Iwijn De Vlaminck, Stephen R. Quake, Hailiang Dong, Brian P. Hedlund, Wesley D. Swingley: Single-Cell-Genomics-Facilitated Read Binning of Candidate Phylum EM19 Genomes from Geothermal Spring Metagenomes. In: Applied and environmental microbiology, Band 82, Nr. 4, 15. Februar 2016, S. 992–1003; doi:10.1128/AEM.03140-15, PMID 26637598, PMC 4751853 (freier Volltext), ResearchGate:285757267, Epub 5. Februar 2016 (englisch). Dazu: Supplement 1 (PDF).
- ↑ NCBI Taxonomy Browser: Candidatus Hydrothermae (Tree), "Candidatus Hydrothermae" Jungbluth et al. 2017, … (phylum).
- ↑ NCBI Taxonomy Browser: pink filament bacterium EM-3 (species), Dazu: Nucleotide: Eubacteria EM 3 16S ribosomal RNA, partiel sequence, GenBank: U05660.1.
- ↑ a b Joseph G. Zeikus, Ralph S. Wolfe: Methanobacterium thermoautotrophicus sp. n., an Anaerobic, Autotrophic, Extreme Thermophile. In: ASM Journals: Journal of Bacteriology, Band 102, Nr. 2, Februar 1972, S. 707–713/715; doi:10.1128/jb.109.2.707-713.1972, PMID 4550816, PMC 285196 (freier Volltext).
- ↑ Kenneth A. Sandbeck, David M. Ward: Temperature Adaptations in the Terminal Processes of Anaerobic Decomposition of Yellowstone National Park and Icelandic Hot Spring Microbial Mats. In: Applied and Environmental Microbiology (AEM), Band 4, Nr. 4, S. 844–851, 1. Oktober 1982; doi:10.1128/aem.44.4.844-851.1982, PMID 16346109, PMC 242107 (freier Volltext), PDF.
- ↑ NCBI Nucleotdie: Sulfolobus sp. (from Octopus Spring 1) ….
- ↑ Luke J. McKay, Olivia D. Nigro, Mensur Dlakić, Karen M. Luttrell, Douglas B. Rusch, Matthew W. Fields, William P. Inskeep: Sulfur cycling and host-virus interactions in Aquificales-dominated biofilms from Yellowstone’s hottest ecosystems. In: Nature: The ISME Journal, Band 16, 14. Oktober 2021, S. 842–855; doi:10.1038/s41396-021-01132-4.
- ↑ Rafael R. de la Haba, André Antunes,, Brian P. Hedlund: Editorial: Extremophiles: Microbial genomics and taxogenomics. In: Frontiers in Microbiology: Sec. Extreme Microbiology, Extremophiles: Microbial Genomics and Taxogenomics, Band 13, 2. August 2022; doi:10.3389/fmicb.2022.984632, PMID 35983330, PMC 9379316 (freier Volltext).

