Phylogenetischer Baum
Ein phylogenetischer Baum ist ein Baum, der die evolutionären Beziehungen zwischen verschiedenen Arten oder anderen Einheiten darstellt, von denen man vermutet, dass sie einen gemeinsamen Vorfahren besitzen. Damit ist ein phylogenetischer Baum eine Form des Kladogramms. In einem phylogenetischen Baum repräsentiert jeder Knoten mit Nachfahren den „nächsten gemeinsamen Verwandten“ dieser Nachfahren. Die Kantenlänge entspricht meist der geschätzten Zeit, in der sich die Arten separiert haben, oder der Anzahl der Mutationen während dieser Entwicklung. Jeder Knoten in einem phylogenetischen Baum wird als „taxonomische Einheit“ bezeichnet, wobei man innere Knoten oft als „hypothetische taxonomische Einheiten“ bezeichnet, wenn die entsprechenden Arten oder Einheiten nicht beobachtet werden können.

1990 wurde von Carl Woese et al. der 3-Domänen-Stammbaum des Lebens vorgestellt (siehe Abb.), der erstmals auf molekular-phylogenetischen Analysen (rRNA-Sequenzvergleichen) basiert.[1] Zwei der drei Lebenslinien (Domänen) bestehen ausschließlich aus Mikroorganismen. Dieses Stammbaumkonzept wurde von Jan Sapp als "The New Foundations of Evolution" bezeichnet.[2] Es wurde seither häufig übernommen und modifiziert.
Datenquellen und Interpretation
[Bearbeiten | Quelltext bearbeiten]Phylogenetische Bäume werden heute meist anhand von sequenzierten Genen der untersuchten Spezies aufgebaut. Dabei berechnet man ein Sequenzalignment des gleichen Gens (oder evtl. der gleichen Gene) dieser Arten und verwendet die im Alignment erscheinenden Ähnlichkeiten und Unterschiede, um den Baum aufzubauen. Arten, deren Sequenzen ähnlich sind, liegen im Baum dann wahrscheinlich näher beieinander als solche mit stark unterschiedlichen Sequenzen. Da die Komplexität der Berechnung solcher Bäume jedoch mit der Anzahl der Sequenzen exponentiell ansteigt, verwendet man Heuristiken, um die Bäume zu generieren. Zu den Standardmethoden der molekular-phylogenetischen Baumkonstruktionsmethoden gehören Maximum-Likelihood-, Neighbor-Joining-, Maximum-Parsimony- und Bayessche Analyse-Methoden.
Ziel der Erstellung phylogenetischer Bäume ist es, die Evolution möglichst detailliert zu rekonstruieren und zu beschreiben. Allerdings weiß man heute, dass die Gene sich nicht gleichmäßig entwickelt haben. Einige Gene, die heute im Menschen vorkommen, haben beispielsweise nur gemeinsame Vorfahren mit dem Schimpansen, andere kommen in allen Säugetieren vor etc.
Deshalb können bei der phylogenetischen Analyse verschiedener Gene der gleichen Spezies unterschiedliche phylogenetische Bäume entstehen, die für sich jedoch alle korrekt sind. Um die Entstehungspunkte und Verzweigungen bei der Evolution der einzelnen Arten festzustellen, müssen deshalb verschiedene Genregionen untersucht werden. Weiterhin sollten Ergebnisse aus der klassischen Phylogenie sowie morphologische Merkmale zur Interpretation hinzugezogen werden.
Um diese Probleme in den Griff zu bekommen, werden neuerdings eine große Anzahl von Genen gleichzeitig untersucht; Unregelmäßigkeiten in der Entwicklungsgeschwindigkeit gleichen sich so aus.
Optimal können die Abstammungsverhältnisse erschlossen werden, wenn von allen betrachteten Spezies das gesamte Genom bekannt und seine Gene bestimmt sind. Nach Zuordnung aller zueinander orthologer Gene bleiben diejenigen Gene übrig, in denen sich die Spezies unterscheiden. Phylogenetische Bäume, die auf solchen Betrachtungen der Orthologie beruhen, gelten als die verlässlichsten und stehen für alle sequenzierten Spezies zur Verfügung, insbesondere die so erstellten Stammbäume der Bakterien und Archäen bieten bereits einen detaillierten Überblick über deren Abstammungsverhältnisse.[3][4]
Wichtige Grundlagenarbeiten zur Konstruktion von phylogenetischen Bäumen wurden Ende der 1960er Jahre von Walter M. Fitch erarbeitet.
Gewurzelte und ungewurzelte Bäume
[Bearbeiten | Quelltext bearbeiten]Ein gewurzelter phylogenetischer Baum ist ein gerichteter Baum mit einer bestimmten einzelnen Kante, in der die Position des nächsten gemeinsamen Vorfahren aller Einheiten im Baum vermutet wird. (Dieser Vorfahre selbst entspräche einem zusätzlichen Knoten.)
Ein ungewurzelter Baum dagegen besitzt keinen ausgezeichneten nächsten gemeinsamen Vorfahren, sondern soll lediglich die Verwandtschaftsnähe oder -ferne der einzelnen Arten darstellen. In der Regel werden ungewurzelte Bäume lediglich als Bearbeitungsschritt eingeführt. Da die tatsächliche Evolution zeitlich gerichtet abgelaufen ist, kann ein ungewurzelter Baum nur ein unvollkommenes Modell der Realität liefern.
Um einen rechnerisch ermittelten Baum zu verwurzeln, sind verschiedene Methoden anwendbar[5]. Die bei weitem bedeutsamste ist die Verwendung von Außengruppen. Eine Außengruppe ist ein mit analysiertes Taxon, das mit der untersuchten Gruppe nahe verwandt ist, aber bekanntermaßen und eindeutig außerhalb des Verwandtschaftskreises steht. Die mit analysierte Sequenz der Außengruppe ermöglicht fast immer die Verwurzelung. Die Methode versagt, wenn die Sequenz der Außengruppe stark von den analysierten Sequenzen abweicht, z. B., weil sie zu entfernt verwandt ist. In diesem Fall entspricht der Vergleich mit ihrer Sequenz mehr oder weniger dem mit einer zufälligen. Andere mögliche Verwurzelungsverfahren, die auf angenommenen Veränderungsraten (molekulare Uhr) oder der Annahme irreversibler Veränderungsmuster beruhen, sind deshalb besonders bei sehr basalen Verzweigungen, für die keine nahe verwandte Außengruppe zur Verfügung steht, wichtig.
Näheres zu gewurzelten und ungewurzelten Bäumen ist im Artikel Baum (Graphentheorie) nachzulesen.
Unterschiede zwischen Gen- und Speziesbäumen
[Bearbeiten | Quelltext bearbeiten]
Verschiedene Formen der Gen/Speziesentwicklung: Üblicherweise wird von dem ersten Fall ausgegangen: Speziation geht mit der Aufspaltung der Genentwicklung einher. Eine Rekonstruktion des Artenbaumes wird erschwert durch die drei anderen Fälle:
- das Gen wird nur von einer der neuen Spezies übernommen (Genverlust)
- das Gen wird dupliziert, was bei einer anschließenden Speziation (nicht gezeigt) zu zweideutigen Vergleichsmöglichkeiten führt
- es findet horizontaler Gentransfer zwischen zwei unterschiedlichen Arten statt, die dadurch bei der Baumrekonstruktion fälschlicherweise zusammengerückt werden
Methoden
[Bearbeiten | Quelltext bearbeiten]Sequenzanalysen
[Bearbeiten | Quelltext bearbeiten]Übliche Methoden der Bioinformatik zur phylogenetischen Sequenzanalyse sind Parsimony, bei der die geringste Anzahl von „Erklärungen“, in diesem Fall Sequenzübereinstimmungen, die Abstammungsverhältnisse klären soll. Beim Neighbour-joining werden alle Sequenzen mit allen in einem Alignment verglichen, die jeweils ähnlichsten zueinander werden als verwandt aufgefasst und in der nächsten Runde des Joinings als eine gemeinsame Art behandelt, bis ein vollständiger Baum entsteht. Gegenwärtig am häufigsten verwendet wird das Maximum-Likelihood-Modell, welches auf statistischen Annahmen über die Evolution von Sequenzen beruht.
Orthologieuntersuchungen
[Bearbeiten | Quelltext bearbeiten]Sind viele Genome bekannt und sind die einzelnen Gene darin charakterisiert, wie dies bereits insbesondere bei den Bakterien der Fall ist, können orthologe Gene zwischen Individuen markiert werden. Alles, was nicht ortholog ist, entstammt einer Insertion oder Deletion eines Gens, je nachdem, welche zeitliche Reihenfolge angenommen wird. Damit müssen nur noch solche Ereignisse analysiert werden, um die Abstammungsverhältnisse festzustellen.[6]
Kritik
[Bearbeiten | Quelltext bearbeiten]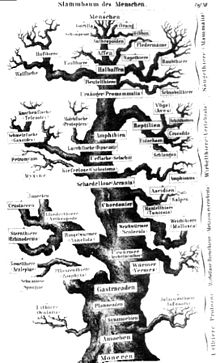
- Per Definition können phylogenetische Bäume Hybridbildung und lateralen Gentransfer, die ebenfalls wichtige Methoden der Genübertragung sind, nicht darstellen. Deshalb vertreten einige Forscher die Ansicht, dass man nicht einen Baum sondern vielmehr ein phylogenetisches Netzwerk aufbauen sollte (das sich im Sinne der Graphentheorie von einem Baum dadurch unterscheidet, dass es „Querverbindungen“ zwischen sonst nicht direkt verwandten Arten zulässt).
- Bäume, die ausgestorbene Arten nicht enthalten, müssen mit Vorsicht interpretiert werden (siehe auch obige Bemerkung zur Interpretation).
Geschichte der Baum-Metapher
[Bearbeiten | Quelltext bearbeiten]
Die Bezeichnung Baum ist abgeleitet von früheren Vorstellungen des Lebens als Fortschritt von „niedrigeren“ zu „höheren“, komplexeren Formen, wobei die jeweils zugeschriebene Entwicklungshöhe durch die Höhe der Platzierung im „evolutionären Stammbaum“ angedeutet wurde. Als Vorlage für die grafische Darstellung eines solchen Stammbaums diente häufig ein realer Baum.
Charles Darwin notierte bereits 1837, dass eine Korallen-Metapher gegenüber dem Baum vorteilhaft wäre. Die lebendigen Anteile säßen auf abgestorbenen Stämmen.[7]
Im Gegensatz zu allen vorherigen Stammbaumkonzepten beruht der 1990 von Carl Woese et al. vorgestellte 3-Domänen-Stammbaum des Lebens zum ersten Mal auf molekular-phylogenetischen Untersuchungsmethoden: "Classical phenotypic criteria are beeing replaced by molecular criteria".[1] Dieser Stammbaum umfasst als Grundlage erstmals auch die mikrobielle Evolution.[2] Offensichtlich war die Geschichte des Lebens ungefähr zu drei Vierteln mikrobiell und das jetzt existierende Leben auf der Erde besteht vorwiegend aus Mikroorganismen.[8]
Eine Veränderung der Stammbaum-Metapher in die eines „Stammflusses“ wurde von Richard Dawkins vorgeschlagen: Man könne das Kladogramm als sich verzweigendes Fluss-System deuten, was u. a. den Vorteil habe, dass diese Metapher keine Höherentwicklung suggeriere.[7]
Siehe auch
[Bearbeiten | Quelltext bearbeiten]Literatur
[Bearbeiten | Quelltext bearbeiten]- Walter M. Fitch, Emanuel Margoliash: Construction of phylogenetic trees. In: Science. Band 155, Nr. 3760, 1967, S. 279–284, doi:10.1126/science.155.3760.279, Volltext (PDF; 356 kB)
- Volker Knoop, Kai Müller: Gene und Stammbäume. Ein Handbuch zur molekularen Phylogenetik. Spektrum Akademischer Verlag, Heidelberg 2006, ISBN 3-8274-1642-6.
- Arndt von Haeseler, Dorit Liebers: Molekulare Evolution. Fischer, Frankfurt am Main 2003, ISBN 3-596-15365-4.
- Bernhard Wiesemüller, Hartmut Rothe, Winfried Henke: Phylogenetische Systematik. Springer, Berlin 2003, ISBN 3-540-43643-X.
- Joseph Felsenstein: Inferring phylogenies. Sinauer Associates, Sunderland, Mass. 2004, ISBN 0-87893-177-5 (580 S.).
- Jan Sapp: The New Foundations of Evolution: On the Tree of Life. Oxford University Press, New York 2009, ISBN 978-0-19-973438-2.
Weblinks
[Bearbeiten | Quelltext bearbeiten]- OneZoom: Tree of Life – Zoombarer Stammbaum aller rezenten Lebewesen-Arten
- Phyletischer Baum von nahezu allen über 4.500 rezenten Säugetierarten, In: Nature. Band 446, 29. März 2007. PMID 17392779
- "Open Tree of Life" (2,3 Millionen Arten)
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ a b c Carl R. Woese, Otto Kandler, Mark L. Wheelis: Towards a natural system of organisms: Proposal for the domains Archaea, Bacteria and Eucarya. In: Proceedings of the National Academy of Sciences. Band 87, Nr. 12, 1990, S. 4576–4579, doi:10.1073/pnas.87.12.4576, PMID 2112744, PMC 54159 (freier Volltext) – (pnas.org [PDF]).
- ↑ a b Jan Sapp: The New Foundations of Evolution: On the Tree of Life. Oxford University Press, New York 2009, ISBN 978-0-19-973438-2 (Google Books [abgerufen am 19. Februar 2024]).
- ↑ OMAbrowser: Orthology Prediction - Algorithm ( vom 3. Mai 2015 im Internet Archive)
- ↑ Status of the OMA Orthologs Project (Enthält aktuelle Bäume)
- ↑ John P. Huelsenbeck, Jonathan P. Bollback, Amy M. Levine: Inferring the Root of a Phylogenetic Tree. In: Systematic Biology. 51(1), 2002, S. 32–43. doi:10.1080/106351502753475862
- ↑ G. Fang, N. Bhardwaj, R. Robilotto, M. B. Gerstein: Getting Started in Gene Orthology and Functional Analysis. In: PLoS Comput Biol. Band 6, Nr. 3, 2010, S. e100073, doi:10.1371/journal.pcbi.1000703 (ploscompbiol.org).
- ↑ a b Wiebke Rögener-Schwarz: Der Stammbaum war einmal. Abgerufen am 8. Februar 2023.
- ↑ Franklin M. Harold: In Search of Cell History: The Evolution of Life's Building Blocks. University of Chicago Press, Chicago, London 2014, ISBN 978-0-226-17428-0, S. 35.